Un gène, un ARN, une protéine ...
Le flux d'information va de l'ADN vers le protéines le plus souvent. L'expérience de Beadle et Tatum en 1941 avait mis en évidence le lien entre le défaut d'un gène et d'une enzyme. On a alors forgé l'expression "un gène -> une enzyme" et plus tard : "un gène -> un ARNm -> une protéine " (on un polypeptide). C'est devenu une de ces phrases faciles à retenir qu'on répète en classe et que les élèves apprécient pour son apparente simplicité.
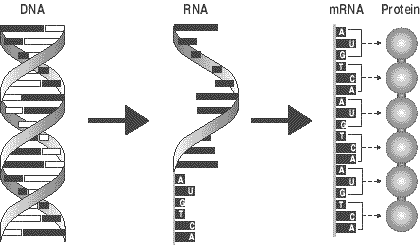 |
| 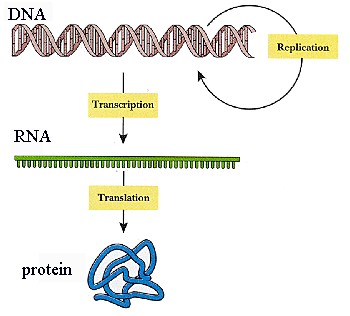
Fig 1 a et b : Un gène -> un ARN , une protéine sources thebody.com et westvalley.edu/svensson
C'était simple, clair et facile a retenir. On rajoute parfois - comme une exception et pour les classes avancées - que l'épissage alternatif peut complexifier ce schéma trop beau pour être vraiment exact.
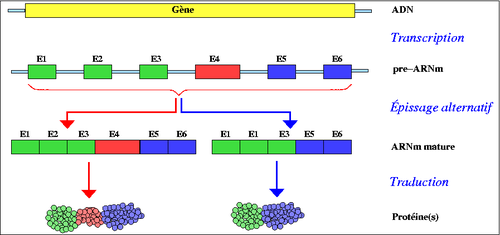
Fig 2 L'épissage alternatif produit plusieurs protéines à partir d'une seul gène : Source : Wikipédia
Un gène, un ARNm, une protéine ... vrai à 6% seulement !
Une étude récente - rapporté par Heidi Ledford dans Nature News en novembre - montre que très rares sont nos gènes qui ne produisent qu'une seule protéine.Pan, Q. et al. (2008) ont converti les ARNm trouvés dans des tissus différents (cerveau, foie, muscle, poumons et d'autres) en ADN et les ont séquencés. Plus de 400 millions de séquences ont ainsi permis d'établir que que 92 - 94% de nos gènes produisent plus d'une protéine. Ils révèlent aussi qu'il y a environ 100,000 évènements de splicing fréquents (épissage) dans nos tissus principaux.
On commence a voir mieux comment le nombre énorme de protéines de nos cellules peut venir de si peu de gènes. Selon le dossier pédagogique de l'expo Chromosome Walk " Les êtres humains ont quelque 21'000 gènes à l’origine de plusieurs centaines de milliers de protéines différentes "Source : Groupe communication de Swiss-Prot UNIGE. (2008).
Peut-on continuer à écrire au tableau "un gène -> un ARNm -> une protéine" comme si de rien n'était ?
Pour simplifier un sujet bien complexe, est-ce tout de même acceptable de présenter ce schéma à des élèves qui ont de la peine à retenir même ces bases simples ?Il n'y a sans doute pas de réponse universelle à cette question. On voit bien la tension entre une vision de l'enseignement qui cherche dans la simplification des concepts une réduction de l'effort d'apprentissage, et une autre vision qui cherche à présenter une biologie dynamique, en mouvement, actualisée qui motive plus les élèves et ... compenserait la complexité accrue.
Faut-il déduire que nous avons enseigné faux à nos élèves ?
Faut-il reconnaitre que nous - ou la science - nous sommes trompés ?
Parler de faux ou reconnaitre qu'on se serait trompé procède d'une vision de la science qui trouverait le "vrai"et où le "scientifiquement prouvé " serait certitude définitive.Si on a enseigné des telles certitudes, il apparait alors qu'on s'est trompé ou que la science s'est trompée.
Et l'enseignant se retrouve dans ce cas avec le dilemme de risquer perdre de sa crédibilité en reconnaissant une erreur ou de la perdre définitivement en cherchant à la cacher le jour ça se découvre. A l'heure d'Internet et de Wikipédia c'est un gros risque...
D'un autre côté, cet exemple montre bien qu'en science il n'y a jamais de certitude définitive, il n'y a que des hypothèses courantes.
On le voit bien ici : ce schéma ADN-> ARN-> protéine n'est pas "faux". Mais oui, il est faux d'imaginer une parfaite correspondance entre un gène et un seul ARN puis un seul polypeptide. Et d'ailleurs aussi d'imaginer que le flux soit toujours unidirectionnel.
Ainsi si cette recherche -et d'autres avant elle- remettent en question ce qu'on a dit autrefois, c'est pour l'affiner, le préciser en délimiter le domaine de validité, nuancer la portée, pas pour le rejeter complètement. Les hypothèses deviennent plus solides au fur et à mesure qu'on les précise et les nuance.
Si on a enseigné une science d'hypothèses courantes : "d'après ce qu'on sait actuellement, voici comment les choses se passent... " on peut alors -sans perdre la face ni dénaturer la science - compléter avec les découvertes nouvelles, ou les préciser et donner une touche d'actualité en prise sur le monde des élèves, qui les motive souvent.
Mettre les élèves dans le rôle de l'expert critique pour leur apprendre à trier ?
Une manière élégante de gérer ces situations où l'on vient d'apprendre que ce qu'on s'apprête à présenter aux élèves n'est plus à jour m'a été racontée par une jeune enseignante (Sabine de Vevey) : après avoir lu le bio-tremplin sur la pseudo-répartition des goûts sur la langue quelques jours avant un TP sur ce sujet, elle a décidé de mettre les élèves dans la position - très scientifique - de juger eux-mêmes et de voir le progrès. Elle a d'abord fait le TP classique : demander aux élèves de produire la carte des sensibilités aux goûts sur la langue. Certains sont arrivés à la carte institutionnalisée (les élèves doubleurs notamment) et d'autres non.Puis elle a montré l'article de Nature (Chandrashekar, et al. 2006) qui révélait que cette carte ne correspond pas aux connaissances actuelles.

Ensuite elle a montré 3-4 ouvrages - très sérieux - qui présentaient une telle carte ... mais chaque fois différente !
1. Marieb, E.N., R. Lachaîne, and L. Moussakova, Anatomie et physiologie humaines. 2000: De Boeck Université.
2.Perilleux, SVT 3ème. 1999: Nathan.
3. Schäffler, A. and N. Menche, Anatomie, physiologie, biologie : abrégé d'enseignement pour les professions de santé. 2004: Maloine.
4. Anselme, B., Repères Pratiques. Le corps humain, Editions Nathan, 1998.
Quelle expérimentation pratique-t-on vraiment ?
Comment on est arrivé des années durant à ce que les élèves produisent une carte "correcte" dans ces condition pose des question sur l'expérimentation qu'on arrive à pratiquer en classe... De Vecchi, G. (2006) analyse d'ailleurs assez bien ces difficultés et incite à plus de modestie : avoir l'ambition d'éprouver une théorie, plutôt que de la démontrer, par exemple.Et comment une pareille différence dans les sources n'a pas attiré l'attention pose bien des questions sur les liens entre les savoirs de référence et ceux qu'on enseigne... Comment ce que la recherche découvre parvient si difficilement jusque dans les classes.
C'est la mission de ces Bio-tremplins d'aider sur ce plan-là...
Intéressant retournement d'un problème en élégante solution non ?
Les élèves ont été très intéressés et fiers d'être partie prenante d'une actualisation des savoirs scolaires. Leur donner juste une carte mise à jour n'aurait pas eu le même effet. Dans ce rôle critique, où ils font face à la complexité et où on les aide à trier ils ont pu construire de solides connaissances sur la base de documents authentiques récents. On sait combien la démotivation des élèves face aux apprentissages en science est nettement liée à leur difficulté à voir des liens avec le monde qu'ils vivent.Et l'estime qu'ils ont de leur enseignante s'en est trouvée accrue, je pense.
Cet exemple met en évidence combien le rôle que les élèves ont effectivement - ici d'analyse critique et de choix dans l'information - est déterminant pour apprendre dans une société de la surabondance d'information.
La biologie est une discipline qui a la chance de pouvoir offrir à ses élèves un visage dynamique, des connaissances toutes fraîches, peut-on en profiter pour intéresser les élèves ?
Sources :
- De Vecchi, G. (2006). Enseigner l'expérimental en classe : pour une véritable éducation scientifique Paris: Hachette éducation.
- Groupe communication du groupe Swiss-Prot. (2008). Chromosome Walk, dossier pedagogique Genève: Institut Suisse de Bioinformatique.
- Ledford, Heidi. (2008). Human genes are multitaskers. Nature News2 November 2008| doi:10.1038/news.2008.1199
- Wang, E. T. et al. Alternative isoform regulation in human tissue transcriptomes Nature doi:10.1038/nature07509 (2008).
- Pan, Q. et al. Deep surveying of alternative splicing complexity in the human transcriptome by high-throughput sequencing Nature Genet. doi:10.1038/ng.259 (2008).
- Jayaram Chandrashekar, Mark A. Hoon, Nicholas J. P. Ryba and Charles S. Zuker Review ArticleThe receptors and cells for mammalian taste Nature 444, 288-294 (16 November 2006) | doi:10.1038/nature05401;


Aucun commentaire:
Enregistrer un commentaire